 Revolutionizing the Clade Determination of Candida auris: The Breakthrough of “CLADE-FINDER”
Revolutionizing the Clade Determination of Candida auris: The Breakthrough of “CLADE-FINDER”
First identified in Japan in 2009, Candida auris has been reported in over 60 countries, causing numerous outbreaks in healthcare settings. C. auris, a multi-drug-resistant fungal pathogen, has rapidly become a global health threat. Declared an “urgent antimicrobial threat” by the CDC in 2019, its ability to colonize environments, resist antifungal treatments, and spread in healthcare environments presents a significant challenge.
C. auris is characterized by considerable genetic diversity, divided into various clades (lineages), which exhibit geographical differences and antifungal resistance profiles. So far, six main clades have been identified, each with unique genetic and phenotypic characteristics. These clades are crucial for understanding the spread of infection and developing targeted treatment strategies.
The Clade Division of Candida auris
The classification of C. auris into clades has revealed significant variability between different populations. The six main clades recognized so far are:
- Clade I: South Asia
- Clade II: East Asia
- Clade III: Africa
- Clade IV: South America
- Clade V: Iran
- Clade VI: Indomalayan (recently discovered)
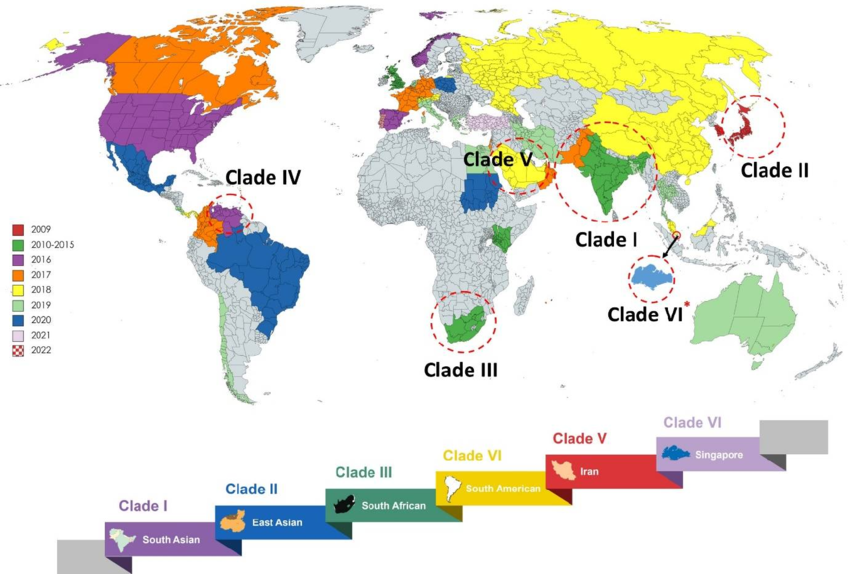
Each clade has unique genetic features that influence its epidemiology and response to antifungal treatments. For example, some clades show greater resistance to certain antifungals, complicating infection management in clinical settings. Determining the clade is therefore essential for epidemiological monitoring and controlling the spread of C. auris, as well as for selecting the most effective antifungal treatment.
Traditional Clade Determination Methods
Traditional clade determination methods, such as whole-genome sequencing (WGS) and short tandem repeat (STR) analysis, are considered the gold standard for identifying C. auris clades. However, these methods are expensive, complex, and require high technical expertise, making them impractical in many healthcare settings, particularly those with limited resources.
Additionally, gene sequencing is time-consuming, a critical factor when dealing with real-time infection outbreaks. Thus, there is a need for alternative methods that are faster, cost-effective, and more efficient for clade determination.
Enter “CLADE-FINDER” a new tool that combines Fourier Transform Infrared Spectroscopy (FTIR) with Artificial Neural Networks (ANN), designed to classify clades in real time, both cost-effectively and efficiently.
Enter “CLADE-FINDER”: An Innovative Tool
To address these challenges, an innovative method called CLADE-FINDER has been developed, combining Fourier-transform infrared (FTIR) spectroscopy with artificial neural networks (ANNs) to classify clades in real-time, economically, and efficiently.
- FTIR spectroscopy is an analytical technique that provides an infrared absorption spectrum, offering a unique biochemical fingerprint for each strain. Although this technique has been widely used for bacterial characterization, its application for fungi has been limited until now. The combination of FTIR with machine learning algorithms, such as ANNs, allows for rapid and precise clade classification of C. auris at a low cost.
- Machine learning, a subset of artificial intelligence, uses algorithms to analyze and interpret large datasets. In this context, machine learning has been employed to develop a predictive model that can quickly identify the clade of C. auris using data obtained through FTIR.
CLADE-FINDER leverages this spectral data, processing it through machine learning algorithms to distinguish between the four main clades of C. auris (I-IV) with remarkable accuracy.
Key Points of the Study
- Dataset Diversity: The study analyzed 74 isolates from hospital outbreaks and global regions. It included representatives from all major clades (I-IV) and an additional clade (V), treated as an outlier.
- High Accuracy: The ANN-based classifier achieved 96% accuracy in clade identification in the validation set, representing a significant advancement in real-time fungal surveillance.
- Rapid Results: Unlike WGS, which takes days, the FTIR-based analysis of CLADE-FINDER produces results in just a few hours, enabling quicker responses to outbreaks.
- Accessibility: This cost-effective approach reduces reliance on specialized equipment and advanced expertise, making it suitable for resource-limited settings.
Implications for Public Health
The accurate and timely classification of C. auris clades has immediate applications:
- Infection Control: Rapid identification of clades can guide isolation strategies and limit transmission in healthcare settings.
- Treatment Guidance: Understanding clade-specific resistance profiles aids in selecting effective antifungal therapies.
- Epidemiological Insights: Monitoring clade distribution enhances understanding of the pathogen’s evolution and spread.
Future Prospects
While CLADE-FINDER represents a significant advancement, further research is needed to include additional clades, such as the recently identified Indomalayan clade (Clade VI). Expanding its applicability to other fungal pathogens could revolutionize fungal diagnostics in general.
In a world where antimicrobial resistance is an increasing concern, innovative tools like CLADE-FINDER offer hope. By combining advanced machine learning techniques with biochemical fingerprints, this study paves the way for smarter, faster, and more accessible microbial surveillance.
 Rivoluzionare la Determinazione dei Cluster Genetici di Candida auris: La Svolta di “CLADE-FINDER”
Rivoluzionare la Determinazione dei Cluster Genetici di Candida auris: La Svolta di “CLADE-FINDER”
Identificato per la prima volta nel 2009 in Giappone, Candida auris è stato segnalato in oltre 60 paesi, causando numerosi focolai di infezioni in strutture sanitarie. C. auris, un patogeno fungino multi-resistente, è rapidamente diventata una minaccia globale per la salute. Dichiarata “minaccia antimicrobica urgente” dal CDC nel 2019, la sua capacità di colonizzare ambienti, resistere ai trattamenti antifungini e diffondersi negli ambienti sanitari rappresenta una sfida significativa.
Candida auris è caratterizzata da una notevole diversità genetica, suddivisa in diversi cladi (lineages, cluster genetici), che presentano differenze geografiche e dei profili di resistenza agli antifungini. Finora sono stati identificati sei cladi principali, ognuno con caratteristiche genetiche e fenotipiche uniche. Questi cluster genetici sono cruciali per comprendere la diffusione dell’infezione e sviluppare strategie di trattamento mirate.
La suddivisione di Candida auris in cladi
La classificazione di C. auris in cluster genetici ha rivelato una notevole variabilità tra le diverse popolazioni. I sei cladi principali riconosciuti finora sono:
- Clade I: Sud Asia
- Clade II: Est Asia
- Clade III: Africa
- Clade IV: Sud America
- Clade V: Iran
- Clade VI: Indomalayan (scoperto recentemente)

Ogni cluster presenta peculiarità genetiche che influenzano la sua epidemiologia e la risposta ai trattamenti antifungini. Per esempio, alcuni mostrano una resistenza maggiore ad alcuni antifungini rispetto ad altri, il che complica la gestione delle infezioni in ambito clinico. La determinazione del cluster genetico è quindi essenziale per un monitoraggio epidemiologico e controllare la diffusione di C. auris, nonché per scegliere il trattamento antifungino più efficace.
Metodi tradizionali di suddivisione dei cladi
I metodi classici di suddivisione dei cladi, come il sequenziamento dell’intero genoma (WGS) e l’analisi degli STR (Short Tandem Repeats), sono considerati lo standard di riferimento per l’identificazione dei cladi di C. auris. Tuttavia, questi metodi sono costosi, complessi e richiedono un’alta competenza tecnica, il che li rende poco pratici in molte strutture sanitarie, soprattutto in quelle con risorse limitate.
Inoltre, il sequenziamento genico richiede molto tempo, un fattore critico quando si trattano focolai di infezione in tempo reale. Pertanto, esiste la necessità di metodi alternativi più rapidi, economici ed efficaci per la suddivisione dei cladi.
Entra in gioco “CLADE-FINDER“, un nuovo strumento che combina la spettroscopia a trasformata di Fourier (Fourier Transform Infrared, FTIR) con reti neurali artificiali (Artificial Neural Networks, ANN), progettato per classificare i cladi in tempo reale, in modo economico ed efficace.
Introduzione a CLADE-FINDER: una soluzione innovativa
Per affrontare queste sfide, è stato sviluppato un metodo innovativo chiamato CLADE-FINDER, che utilizza la spettroscopia a trasformata di Fourier (FTIR) combinata con l’apprendimento automatico (machine learning).
- La spettroscopia FTIR è una tecnica analitica che fornisce uno spettro di assorbimento infrarosso, un’impronta biochimica unica per ogni ceppo. Sebbene questa tecnica sia stata ampiamente utilizzata per la caratterizzazione batterica, la sua applicazione per i funghi è stata limitata fino ad oggi. La combinazione di FTIR con algoritmi di apprendimento automatico, come le reti neurali artificiali (ANN), consente di classificare rapidamente i cladi di C. auris in modo preciso e a basso costo.
- Il machine learning è un sottoinsieme dell’intelligenza artificiale che utilizza algoritmi per analizzare e interpretare grandi quantità di dati. In questo contesto, l’apprendimento automatico è stato utilizzato per sviluppare un modello predittivo che può identificare rapidamente il clade di C. auris, utilizzando i dati ottenuti tramite FTIR.
CLADE-FINDER sfrutta questi dati spettrali, elaborandoli tramite algoritmi di apprendimento automatico, per distinguere tra i quattro principali cladi di C. auris (I-IV) con un’accuratezza straordinaria.
Punti Chiave dello Studio
- Diversità del Dataset: Lo studio ha analizzato 74 isolati provenienti da focolai ospedalieri e regioni globali. Sono stati inclusi rappresentanti di tutti i principali cladi (I-IV) e un clado aggiuntivo (V), trattato come outlier.
- Alta Accuratezza: Il classificatore basato su ANN ha raggiunto un’accuratezza del 96% nell’identificazione dei cladi nel set di validazione, rappresentando un significativo passo avanti nella sorveglianza fungina in tempo reale.
- Tempi Rapidi: A differenza del WGS, che richiede giorni, l’analisi basata su FTIR di CLADE-FINDER produce risultati in poche ore, consentendo risposte più rapide ai focolai.
- Accessibilità: Questo approccio economico riduce la dipendenza da attrezzature specializzate e competenze avanzate, rendendolo adatto a contesti con risorse limitate.
Implicazioni per la Salute Pubblica
La classificazione accurata e tempestiva dei cluster genetici di C. auris ha applicazioni immediate:
- Controllo delle Infezioni: L’identificazione rapida dei cluster genetici può guidare strategie di isolamento e limitare la trasmissione negli ambienti sanitari.
- Guida al Trattamento: La comprensione dei profili di resistenza specifici per cluster genetico aiuta a selezionare terapie antifungine efficaci.
- Approfondimenti Epidemiologici: Monitorare la distribuzione dei cladi migliora la comprensione dell’evoluzione e della diffusione del patogeno.
Prospettive Future
Sebbene CLADE-FINDER rappresenti un importante progresso, sono necessarie ulteriori ricerche per includere cladi aggiuntivi, come il recentemente identificato clado Indomalayano (clado VI). Espandere la sua applicabilità ad altri patogeni fungini potrebbe rivoluzionare la diagnostica fungina in generale.
In un mondo in cui la resistenza antimicrobica è una preoccupazione crescente, strumenti innovativi come CLADE-FINDER offrono speranza. Combinando tecniche avanzate di apprendimento automatico con impronte biochimiche, questo studio apre la strada a una sorveglianza microbica più intelligente, veloce e accessibile.
Author: Stefano Michienzi
Sources:
“CLADE-FINDER”: Candida auris Lineage Analysis Determination by Fourier Transform Infrared Spectroscopy and Artificial Neural Networks.
Magrì C, De Carolis E, Ivagnes V, Di Pilato V, Spruijtenburg B, Marchese A, Meijer EFJ, Chowdhary A, Sanguinetti M.
Microorganisms. 2024 Oct 26;12(11):2153. doi: 10.3390/microorganisms12112153.
PMID: 39597542
Current Perspectives of Antifungal Therapy: A Special Focus on Candida auris.
Ganeshkumar A, Muthuselvam M, Lima PMN, Rajaram R, Junqueira JC.
J Fungi (Basel). 2024 Jun 6;10(6):408. doi: 10.3390/jof10060408.
PMID: 38921394



